Das vierdimensionale menschliche Genom ist eine Herausforderung für alle naturalistischen Erklärungen
Published: 6 October 2016 (GMT+10)

Das menschliche Erbgut bzw. Genom ist das komplexeste Computer-Betriebssystem im gesamten bekannten Universum. Es steuert superkomplexe biochemische Prozesse so präzise, dass einzelne Moleküle angesprochen werden. Es kontrolliert ein Netzwerk von hunderttausenden Proteinen, die alle miteinander in Wechselwirkung stehen. Es ist ein wunderbares Zeugnis für die kreative Genialität Gottes, und ein exzellentes Beispiel für die wissenschaftliche Bankrott-Erklärung der Neo-Darwinistischen Theorie. Warum? Weil die Evolutionstheorie umso weniger vertreten werden kann, desto komplexer das Leben ist. An superkomplexen Maschinen kann man nicht auf gut Glück herumbasteln, und wenn man es doch versucht, werden sie kaputt gehen. Superkomplexe Maschinen entstehen auch nicht durch zufällige Veränderungen.
Ich meine es ernst, wenn ich das Genom mit einem Betriebssystem eines Computers vergleiche. Das einzige Problem mit dieser Analogie ist, dass es keine Computer gibt, die hinsichtlich ihrer Komplexität oder Effizienz mit einem Genom vergleichbar wären. Die Analogie funktioniert nur, wenn man sozusagen auf der obersten Ebene bleibt, aber das ist ja gerade die Stärke des Vergleichs. Nachdem wir Millionen Stunden lang Computerprogramme geschrieben und debugged haben, ist es uns gerade mal gelungen, Betriebssysteme zu entwickeln, die einen Laptop oder einen Server betreiben können, und trotzdem stürzen sie oft ab. Das menschliche Genom hingegen betreibt eine hyperkomplexe Maschine, nämlich den menschlichen Körper. Die Art und Weise, wie beide Betriebssysteme aufgebaut sind, ist ebenfalls radikal unterschiedlich. Ein Team von Informatikern, Biophysikern und Experten in Bioinformatik (mit anderen Worten, wirklich kluge Leute) hat das Genom des oft unterschätzten E. Coli Bakteriums mit dem Computer-Betriebssystem Linux verglichen (Abbildung 1); dabei haben sie herausgefunden, dass unsere menschengemachten Betriebssysteme viel ineffizienter sind, weil sie zu „kopflastig“ sind.1 Es stellt sich heraus, dass das Genom des Bakteriums nur wenige Instruktionen auf oberster Ebene hat, die einige Prozesse auf mittlerer Ebene steuern, die ihrerseits wieder eine riesige Anzahl von Protein-codierenden Genen steuern. Bei Linux ist es gerade anders herum: Linux konzentriert sich auf die oberste Ebene, und ist daher viel ineffizienter, wenn es darum geht, seine Funktionen zu erfüllen. Das Bakterium kann mit weniger Steuerprozessen wesentlich mehr leisten. Meine Vorhersage ist, dass das Studium des Erbguts die Entwicklung zukünftiger Computer beeinflussen wird.
Unsere Computer arbeiten mit verhältnismäßig einfachen Programmen. Die Programmierer reden hier von „Programmzeilen“. Wir haben alle im Mathematikunterricht gelernt, dass eine Zeile ein eindimensionales Objekt ist. D. h. unsere Computerprogramme sind im Wesentlichen eindimensional. Das menschliche Genom arbeitet hingegen in vier Dimensionen! Das ist eines der größten Zeugnisse für die brillante Kreativität Gottes, das wir haben.
Die Erste Dimension: das DNS Molekül
Das menschliche Genom ist 1,8 m lang und hat in einem einzigen Zellkern Platz. Um einen Vergleich zu geben: Wenn die DNS so dick wie ein menschliches Haar wäre, wäre die DNS mehr als 50 Kilometer lang, und zu einem Knäuel zusammengepresst, das in etwa die Größe eines Golfballs hätte. Bereits jetzt muss uns aufgehen, dass Gott ein unglaublicher Ingenieur ist.
Wenn wir uns die Abfolge von „Buchstaben“ in der DNS anschauen würden, könnte sie wie folgt aussehen:
CTAACCCTAACCCTAACCCTAACCCTAACCCTAACCCTCTGAAAGTGGACCTATCAGCAGGATGTGGGTGGGAGCAGATTAGAGAATA AAAGCAGACTGCCTGAGCCAGCAGTGGCAACCCAATGGGGTCCCTTTCCATACTGTGGAAGCTTCGTTCTTTCACTCTTTGCAATAAAT CTTGCTATTGCTCACTCTTTGGGTCCACACTGCCTTTATGAGCTGTGACACTCACCGCAAAGGTCTGCAGCTTCACTCCTGAGCCAGTG AGACCACAACCCCACCAGAAAGAAGAAACTCAGAACACATCTGAACATCAGAAGAAACAAACTCCGGACGCGCCACCTTTAAGAACTG TAACACTCACCGCGAGGTTCCGCGTCTTCATTCTTGAAGTCAGTGAGACCAAGAACCCACCAATTCCAGACACACTAGGACCCTGAGA CAACCCCTAGAAGAGCACCTGGTTGATAACCCAGTTCCCATCTGGGATTTAGGGGACCTGGACAGCCCGGAAAATGAGCTCCTCATCT CTAACCCAGTTCCCCTGTGGGGATTTAGGGGACCAGGGACAGCCCGTTGCATGAGCCCCTGGACTCTAACCCAGTTCCCTTCTGGAAT TTAGGGGCCCTGGGACAGCCCTGTACATGAGCTCCTGGTCTGTAACACAGTTCCCCTGTGGGGATTTAGGGACTTGGGCCTT
Das sind die ersten 700 „Buchstaben“ des menschlichen Y-Chromosoms. Nicht sehr beeindruckend, nicht wahr? Wenn wir aber dieselbe Sequenz nehmen und die vier „Buchstaben“ mit vier unterschiedlich eingefärbten Pixeln ersetzen, kommt so etwas wie in Abbildung 2 heraus.

Die erste Dimension des Genoms ergibt sich einfach aus der Anordnung der „Buchstaben“. Die „Buchstaben“ kodieren Gene, und diese Gene teilen der Zelle mit, bestimmte Anweisungen auszuführen. Das ist nicht wirklich so kompliziert, aber dabei bleibt es eben nicht.
Die Zweite Dimension: das interaktive Netzwerk
Die zweite Dimension des Genoms hat damit zu tun, wie ein Abschnitt der DNS mit einem anderen Abschnitt in Wechselwirkung steht. Wie wir bereits gesehen haben, ist es recht einfach, die erste Dimension darzustellen. Wenn wir aber versuchen wollen, die zweite Dimension darzustellen, müssten wir zuerst viele Verbindungspfeile einzeichnen, die verschiedene Abschnitte auf der linearen DNS-Kette miteinander verbinden. Es wäre unmöglich, das gesamte interaktive Netzwerk des Genoms darzustellen, und daher muss uns ein kleines Beispiel genügen. Mikro-RNS (miRNS) besteht aus sehr kleinen Molekülen (aus ungefähr 22 Nukleotiden, Grundbausteinen der DNS, Anm. d. Übersetzers), die bei der Steuerung von genetischen Funktionen beteiligt ist. Abbildung 3 zeigt einen Teil des miRNS-Netzwerks, bei dem 13 Gene beteiligt sind, die „hinauf“geregelt werden, um Arteriosklerose (eine Verhärtung der Arterien) zu bewirken. Diese Gene werden von 262 verschiedenen miRNS Molekülen angesprochen, was zu 372 „regelungstechnischen Beziehungen“ führt. Nicht gezeigt sind in Abbildung 3 die 33 anderen Gene, die mit Hilfe von 295 miRNS-Molekülen „herunter“geregelt werden, wenn der Körper Arteriosklerose verhindern will. Denken wir daran, dass dies nur ein kleiner Ausschnitt aus der zweiten Dimension des Genoms ist!
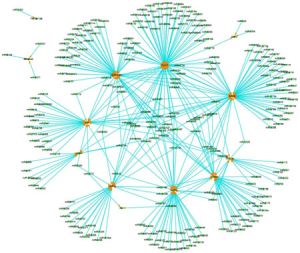
In der zweiten Dimension begegnen uns Spezifitätsfaktoren [Faktoren, die spezifisch bzw. selektiv wirken, Anm. d. Übersetzers], Verstärker, Hemmer, Beschleuniger und Transkriptionsfaktoren [Faktoren, die die Geschwindigkeit des DNS-Kopiervorgangs steuern, Anm. d. Übersetzers]. Das sind alles Proteine, die in die DNS hineincodiert sind, und die sich nach ihrer Herstellung zu einer anderen Stelle des Genoms bewegen, um dort etwas ein- oder auszuschalten. Aber es spielen sich in dieser Dimension noch zusätzliche Dinge ab. Im Prozess der Proteinherstellung „liest“ die Zelle z. B. ein Gen in einem Prozess, den man Transkription nennt, aus. Dabei wird die DNS in ein Molekül namens RNS kopiert – wir haben eine hervorragende Animation dieses Prozesses auf unserer Multimedia-Internetseite. Die RNS kann nun in einem Prozess, den man post-transkriptionelle Regulation nennt, durch andere Faktoren (wie z. B. die miRNS), die an anderen Stellen im Genom codiert sind, aktiviert oder deaktiviert werden.
Das gigantische, viele Millionen Dollar teure Project ENCODE hat uns etwas über das Genom enthüllt, dessen Bedeutung wir immer noch versuchen zu verstehen. Eines der größten Geheimnisse ist nämlich, wie es nur rund 22.000 Gene schaffen, mehr als 300.000 verschiedene Proteine herzustellen. Die Antwort ist, dass die Zelle einen Prozess durchläuft, den man alternatives Spleißen nennt, in dem die Gene in Bruchstücke zerlegt werden, die dann von unterschiedlichen Zellen zu unterschiedlichen Zeiten und unter unterschiedlichen Bedingungen die vielen unterschiedlichen Proteine herstellen. Dieser unglaublich komplexe Vorgang stellt nur einen Teil der zweiten Dimension des Genoms dar.
Die Dritte Dimension: 3D DNS-Architektur
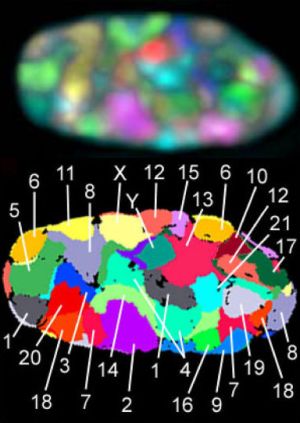
Die dritte Dimension dreht sich darum, welchen Einfluss das Aussehen [gemeint ist die dreidimensionale bzw. 3D-Struktur, Anm. d. Übersetzers] des DNS Moleküls auf die Steuerung und die Wirkung verschiedener Gene hat. Hier haben wir gelernt, dass bestimmte Abschnitte auf der DNS, die tief verborgen in der aufgewickelten DNS-Struktur sind, nicht leicht aktiviert werden können.4 Anders ausgedrückt: Gene, die oft benötigt werden, sind in der Regel leicht zugänglich. Als Gott daher die Information in dem eindimensionalen Genom-Strang niederschrieb, stellte er ganz bewusst eine bestimmte Reihenfolge her, so dass sich die Information am richtigen Platz befindet, wenn die DNS in ihre dreidimensionale Form gefaltet wird.
Eines der großen Aha-Erlebnisse im Humangenomprojekt war, dass Gene, die zusammenwirken, nicht notwendigerweise nebeneinander auf dem Genom liegen müssen. Zu Beginn kursierten Behauptungen wie „Es ist einfach Schrott“ und „Das Genom ist nichts weiter als eine Millionen Jahre alte Ansammlung von genetischen Unfällen“. Doch diese Behauptungen hielten sich nicht lange, als man nämlich anfing zu untersuchen, wie das Genom innerhalb des Zellkerns organisiert ist.5 Es wurde schnell klar, dass nicht nur jedes Chromosom seinen speziellen Platz im Zellkern hat, sondern auch, dass Gene, die zusammenwirken, im Allgemeinen im dreidimensionalen Raum nebeneinander liegen, und zwar sogar dann, wenn sie auf verschiedenen Chromosomen sitzen!
Die Vierte Dimension: Veränderungen der ersten drei Dimensionen
Die vierte Dimension des Genoms hat damit zu tun, wie sich die ersten drei Dimensionen mit der Zeit verändern. Ja, sie haben sich nicht verlesen: Das Aussehen (die dritte Dimension), das interaktive Netzwerk (zweite Dimension) und die Sequenz der „Buchstaben“ (erste Dimension) ändern sich. Das übersteigt so weit die Fähigkeiten selbst unserer modernsten Computer, dass es nicht einmal mehr gerechtfertigt ist, überhaupt einen solchen Vergleich anzustellen.
Die vierte Dimension kann auf unterschiedliche Weise veranschaulicht werden. Beispielsweise wissen wir, dass unterschiedliche Leberzellen eine unterschiedliche Anzahl von Chromosomen enthalten.6 Das ist so, weil die Leber jede Menge Kopien bestimmter Gene braucht, die für den Stoffwechsel und die Entgiftung benötigt werden. Anstatt nun das Genom mit vielen Kopien dieser Gene anzufüllen, macht sich die Leber einfach Kopien für ihre eigenen Zwecke. Ein anderes Beispiel betrifft unser Gehirn, wo bestimmte Zellen eine unterschiedliche Anzahl und Position sogenannter Transposons aufweisen [Transposons oder „springende Gene“ sind Gene, die ihre Position im Genom ändern können, Anm. d. Übersetzers].7 Diese „springenden Gene“ sollen laut den Vertretern der Evolutionstheorie angeblich Überbleibsel von früheren viralen Infektionen sein. Das Problem ist nur: sie sind lebensnotwendig für die Entwicklung des menschlichen Gehirns. Haben Sie das mitbekommen? Das Genom programmiert sich selbst dynamisch um! Das ist etwas, womit sich die Informatiker schon lange beschäftigen: Wie kann man einen sich selbst modifizierenden Code erstellen, der nicht außer Kontrolle gerät? Wir wissen auch, dass Transposons von kritischer Bedeutung bei der Steuerung der embryonalen Entwicklung von Mäusen sind.8 So viel also zur sogenannten „Schrott-DNS“.
Schlussfolgerung
Das Genom ist ein multidimensionales Betriebssystem für einen ultrakomplexen biologischen Computer, mit eingebauter Fehlerkorrektur und sich selbst modifizierendem Code. Es hat eine Vielzahl sich überlappender DNS-, RNS- und struktureller Codes, sowie spezielle DNS- und RNS-Gene. Das Genom wurde bewusst mit jeder Menge Redundanz [Mechanismen zur Verhinderung von Informationsverlust, Anm. d. Übersetzers] entworfen, und zwar von einem hochintelligenten Wesen, das dabei solide technische Prinzipien anwandte. Trotz seiner Redundanz weist das Genom einen erstaunlichen Grad an Kompaktheit auf, der es ermöglicht, aus nur rund 22.000 Protein-codierenden Genen kombinatorisch mehrere hunderttausend verschiedene Proteine herzustellen.
Ich habe eine Herausforderung für die Vertreter der Evolutionstheorie: Erklären sie den Ursprung des Genoms! Charles Darwin schrieb in seinem Buch „Über die Entstehung der Arten“:
„Ließe sich das Vorhandensein eines zusammengesetzten Organs nachweisen, das nicht durch zahlreiche aufeinanderfolgende geringe Abänderungen entstehen könnte, so müsste meine Theorie unbedingt zusammenbrechen.“
Ich weiß, dass dieses Zitat missbraucht wurde (von beiden Seiten der Debatte), aber denken wir einmal für einen Moment darüber nach. Je weniger komplex das Leben ist, desto leichter kann man es mit Hilfe der Darwin´schen Evolutionstheorie erklären. Je komplexer das Leben andererseits ist, umso größer wird das Problem für die Evolutionstheorie. Wir haben gerade gelernt, dass das Genom alles andere als simpel aufgebaut ist. Das sollte alle Vertreter der Evolutionstheorie unangenehm berühren.
Ich behaupte, dass das Genom nicht durch bekannte naturalistische Prozesse entstehen konnte. Der Vertreter der Evolutionstheorie, der sich dieser Herausforderung stellt, muss uns ein funktionierendes Szenario zur Verfügung stellen. Dazu gehört eine Erklärung für die Änderung des Informationsgehalts, sowie eine Beschreibung für die notwendigen Mutationen und selektiv wirkenden Kräfte – und das alles in einem geeigneten Zeitrahmen. Dabei wird sich herausstellen, dass Evolution nicht in der Lage ist, das zu tun, was notwendig wäre, auch nicht in Millionen Jahren.
Literaturangaben
- Yan, K.-K., et al., Comparing genomes to computer operating systems in terms of the topology and evolution of their regulatory control networks. PNAS107(20):9186-9191, 2010. Zurück zum Text.
- Seaman, J., and Sanford, J., Skittle: a 2-dimensional genome visualization tool. BMC Bioinformatics 10:452, 2009. Zurück zum Text.
- Lin, M., Zhao, W., and Weng, J., Dissecting the mechanism of carotid atherosclerosis from the perspective of regulation, International Journal of Molecular Medicine34:1458-1466, 2014. Zurück zum Text.
- van Berkum, N.L., Hi-C: a method to study the three-dimensional architecture of genomes, Journal of Visualized Experiments 6(39):1869, 2010. Zurück zum Text.
- Bolzer, A., et al., Three-dimensional maps of all chromosomes in human male fibroblast nuclei and prometaphase rosettes, PLoS Biol 3(5):e157, 2005. Zurück zum Text.
- Duncan, A.W., et al., The ploidy conveyor of mature hepatocytes as a source of genetic variation, Nature 467:707-710, 2010. Zurück zum Text.
- Baillie, J.K., et al., Somatic retrotransposition alters the genetic landscape of the human brain, Nature 479:534-537, 2011. Zurück zum Text.
- Tomkins, J., 2012. Transposable Elements Key in Embryo Development; icr.org/article/6928, July 25, 2012. Zurück zum Text.


Readers’ comments
Comments are automatically closed 14 days after publication.